植物基因组学研究为植物基因功能、群体遗传、进化和育种研究提供了重要基因组数据资源。近日,浙江大学樊龙江教授课题组在国际知名期刊《Nature Plants》发表了题为“Technology-enabled great leap in deciphering plant genomes”文章,系统收集并分析了自2000年(第一个植物基因组发表)以来测序组装完成的高质量植物基因组,合计包括来自1,575个物种的3,517个基因组。这些测序完成的基因组中,2/3的基因组(2,373个)和1/2的植物物种(793个)是在最近三年(2021-2023)完成的,相比于前20年(2000-2020)呈现出了一个巨大飞跃(图1)。该研究系统分析了完成这些基因组的测序技术和组装算法及其变迁。测序和拼接技术的进步推进了近期植物基因组学研究的快速发展。为了更全面地展示测序物种信息,并提供有关测序技术和组装算法应用情况,他们搭建了N3数据库(N3: plants, genomes, technologies),提供了现有3,517个植物基因组的详细信息,包括测序平台、组装质量、组装工具、可用基因组及其注释文件的下载链接等。该数据库为植物基因组学研究提供了重要资源和支撑。
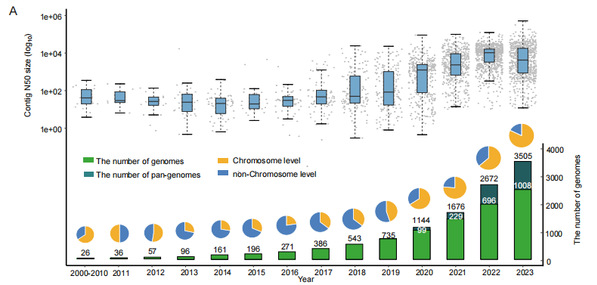
图1 植物基因组测序拼接和组装质量情况
近三年来,植物基因组的组装质量迅速提高,拼接达到染色体水平的基因组比例从前20年的47.3%增长为近三年的73.2%,平均contig N50大小从1.44 Mb增长到11.92 Mb。近三年组装的2,373个基因组涵盖了植物界物种的主要分支(目),同时大量研究致力于更高质量基因组的组装,例如单倍型基因组,泛基因组和端粒到端粒(T2T)基因组(图2)。

图2 植物系统发育及其各主要分支(目)基因组测序物种数量及其相关拼接质量指标。红色表示该目物种为最近三年内才被测定,灰色表示该分支内尚无物种被测序。
在近三年组装的基因组中,94.0%的基因组均利用了三代测序(TGS)技术,已占据主导地位,6.0%的基因组仅使用二代测序(NGS)数据进行拼接。其中三代HiFi数据在2022年的使用比例激增,2023年已达到35.1%。组装算法的创新也为获得更完整的复杂基因组提供了机会。文章详细分析了组装三个阶段的不同特点,统计分析了每个阶段最常使用的软件并详细阐述了其算法的迭代过程。例如基因组拼接步骤,其算法最初是基于测序读序重叠区联配延伸的OLC算法为主,NGS数据出现后德布鲁因图(de Bruijn graph)算法成为主流算法(如SOAPdenovo和Velvet),而随着TGS数据的出现,由于测序读序变长,OLC算法(如Canu)重新换发活力,同时串图(string graph)算法(Hifiasm,Falcon和NextDenovo)可以利用长读序优势,同样成为主流算法。
该研究搭建的N3数据库(http://ibi.zju.edu.cn/N3database/),提供了1,777篇植物基因组相关论文的元数据,涵盖来自1,575个物种的3,517个植物基因组的详细信息。N3数据库提供了代表性物种基因组及其基因注释集,BLAST搜索和JBrowse基因组浏览等功能,为广大研究人员提供了一个及时跟踪获取已测序的植物基因组详细信息的综合平台。
浙江大学农业与生物技术学院和海南研究院博士生谢玲娟、硕士生龚晓娇为论文共同第一作者,樊龙江教授为通讯作者。项目研究得到了浙江省科技厅和海南省科技厅的支持。樊龙江教授团队长期在植物基因组及其演化和环境适应方面开展研究,近年来在植物基因组和泛基因组方面取得了系列成果,分别在Nature Ecology & Evolution、PNAS等刊物上发表相关论文。
论文网址:https://www.nature.com/articles/s41477-024-01655-6
DOI:10.1038/s41477-024-01655-6

